가우스 프로세스 잠재 변수 모델(GPLVM)은 (잠재적으로) 고차원 데이터의 저차원 표현을 학습하기 위해 가우스 프로세스를 사용하는 차원 감소 방법입니다.
입력과 출력이 제공되는 가우스 프로세스 회귀의 일반적인 설정에서 커널을 선택하고 에서 저차원 매핑을 가장 잘 설명하는 하이퍼 매개 변수를 학습합니다.
GPLVM에서 X는 주어지지 않고 y만 주어집니다. 그래서 우리는 커널 하이퍼 파라미터와 함께 배워야 합니다.
X에 대해서 최대 가능성 추론을 하지 않습니다. 대신, Pyro는 가우시안 사전 분포를 설정을 하고 대략적인 (가우스) 사후 평균(q(X|y)과 분산을 학습합니다.
이 글에서는 pyro.contrib.gp 모듈을 사용하여 이 작업을 수행하는 방법을 보여 줍니다.
참고 자료
2022.08.21 - [분석 Python/Pyro] - [Pyro] Application - 1. Bayesian Regression 이해하기
2022.08.28 - [분석 Python/Pyro] - [Pyro] Application - 2. Bayesian Regression 이해하기 2
2022.08.28 - [분석 Python/Pyro] - [Pyro] Application - 3. Gaussian Process 이해하기
2022.08.29 - [분석 Python/Pyro] - [Pyro] Application - 5. GP Bayesian Optimization
Import Library
import os
import matplotlib.pyplot as plt
import pandas as pd
import torch
from torch.nn import Parameter
import pyro
import pyro.contrib.gp as gp
import pyro.distributions as dist
import pyro.ops.stats as stats
smoke_test = ('CI' in os.environ) # ignore; used to check code integrity in the Pyro repo
assert pyro.__version__.startswith('1.8.1')
pyro.set_rng_seed(1)Dataset
사용할 데이터는 mice로부터 얻은 48개 유전자에 대한 단세포 qPCR 데이터로 구성됩니다.
이 데이터는 Open Data Science 저장소에서 사용할 수 있습니다. 데이터에는 48개의 열이 포함되어 있으며 각 열은 각 유전자의 (정규화된) 측정값에 해당합니다.
세포는 발달하는 동안 분화되어 이러한 데이터는 발달의 다양한 단계에서 획득되었습니다. 1셀 단계부터 64셀 단계까지 다양한 단계가 표시됩니다. 32 세포 단계의 경우, 데이터는 '트로펙토더미'(TE)와 '내 세포질량'(ICM)으로 더 분화되며, ICM은 64 세포 단계에서 '에피 블라스트'(Epiblast)와 '원시 내피'(PE)로 더 분화됩니다.
데이터 세트의 각 행에는 다음 단계 중 하나가 레이블링됩니다.
URL = "https://raw.githubusercontent.com/sods/ods/master/datasets/guo_qpcr.csv"
df = pd.read_csv(URL, index_col=0)
print("Data shape: {}\n{}\n".format(df.shape, "-" * 21))
print("Data labels: {}\n{}\n".format(df.index.unique().tolist(), "-" * 86))
print("Show a small subset of the data:")
df.head()
Modelling
먼저 출력 텐서를 정의해야 합니다.
모든 유전자의 값을 예측하기 위해서는 Gaussian Process가 필요합니다.
따라서 필요한 모양은 num_GPS x num_data = 48 x 437입니다.
여기서 y에 대해서 잠재 변수를 찾는 모델링을 하는 것이다.
data = torch.tensor(df.values, dtype=torch.get_default_dtype())
# we need to transpose data to correct its shape
y = data.t()
이제 가장 흥미로운 부분이 나옵니다.
Pyro는 관찰된 데이터가 잠재된 구조를 가지고 있다는 것을 알고 있습니다.
특히 서로 다른 데이터 포인트는 서로 다른 세포 단계에 해당합니다.
GPLVM이 감독되지 않은 방식으로 이 구조를 배우기를 원합니다.
원칙적으로, Pyro가 추론을 잘한다면, Pyro는 적어도 합리적인 사전(priors)을 선택한다면, 이 구조를 발견할 수 있을 것입니다.
먼저, Pyro는 잠재 공간의 차원(the dimension of our latent space)을 선택해야 합니다.
Pyro는 모델이 셀 분기 유형(TE, ICM, PE, EPI)에서 '캡처 시간'( 1,2,4,8,16,32,64)을 분리하기를 원하기 때문에 선택합니다.
다음으로, 이전 X에 대한 prior 평균을 설정할 때 첫 번째 차원을 관찰된 캡처 시간과 동일하게 설정합니다.
이것은 GPLVM이 관심 있는 구조를 발견하는 데 도움이 될 것이고 해석하기 더 쉬운 방식으로 그 구조가 축 정렬될 가능성이 더 높아지게 할 것입니다.
capture_time = y.new_tensor([int(cell_name.split(" ")[0]) for cell_name in df.index.values])
# we scale the time into the interval [0, 1]
time = capture_time.log2() / 6
# we setup the mean of our prior over X
X_prior_mean = torch.zeros(y.size(1), 2) # shape: 437 x 2
X_prior_mean[:, 0] = time훈련을 더 빠르게 하기 위해 sparse gaussian process 추론을 사용할 것입니다.
또한 X를 매개변수로 정의해야 사전 및 가이드(변동 분포)를 설정할 수 있습니다.
kernel = gp.kernels.RBF(input_dim=2, lengthscale=torch.ones(2))
# we clone here so that we don't change our prior during the course of training
X = Parameter(X_prior_mean.clone())
# we will use SparseGPRegression model with num_inducing=32;
# initial values for Xu are sampled randomly from X_prior_mean
Xu = stats.resample(X_prior_mean.clone(), 32)
gplvm = gp.models.SparseGPRegression(X, y, kernel, Xu, noise=torch.tensor(0.01), jitter=1e-5)
`. to_event()`를 사용하여 X에 대한 사전 분포에 batch_shape가 없음을 Pyro에 알립니다
Parameterized 클래스의 autoguide() 메서드를 사용하여 X에 대한 자동 Normal 가이드를 설정합니다.
# we use `.to_event()` to tell Pyro that the prior distribution for X has no batch_shape
gplvm.X = pyro.nn.PyroSample(dist.Normal(X_prior_mean, 0.1).to_event())
gplvm.autoguide("X", dist.Normal)Inference
가우스 프로세스 튜토리얼에서 언급한 대로 도우미 함수 gp.util.train을 사용하여 Pyro GP 모듈을 교육할 수 있습니다. 기본적으로 이 도우미 기능은 학습률이 0.01인 Adam 옵티마이저를 사용합니다.
# note that training is expected to take a minute or so
losses = gp.util.train(gplvm, num_steps=4000)
# let's plot the loss curve after 4000 steps of training
plt.plot(losses)
plt.show()
추론 후에, 추정된 분포 q(X) ~ P(X|Y)의 평균과 편차를 파라미터 X_LOC , X_SCALE에 저장됩니다.
q(X)로 부터 샘플을 얻기 위해서는 gplvm의 mode를 가이드로 바꿔야 합니다.
gplvm.mode = "guide"
X = gplvm.X # draw a sample from the guide of the variable XVisualizing the result
여기선 학습된 것을 기반으로 시각화를 하면 다음과 같습니다.
각 INDEX에 대해서 학습된 파라미터로 시각화를 하게 되면, 나뉘는 형태를 잘 학습한 것을 알 수 있습니다.
plt.figure(figsize=(8, 6))
colors = plt.get_cmap("tab10").colors[::-1]
labels = df.index.unique()
X = gplvm.X_loc.detach().numpy()
for i, label in enumerate(labels):
X_i = X[df.index == label]
plt.scatter(X_i[:, 0], X_i[:, 1], c=[colors[i]], label=label)
plt.legend()
plt.xlabel("pseudotime", fontsize=14)
plt.ylabel("branching", fontsize=14)
plt.title("GPLVM on Single-Cell qPCR data", fontsize=16)
plt.show()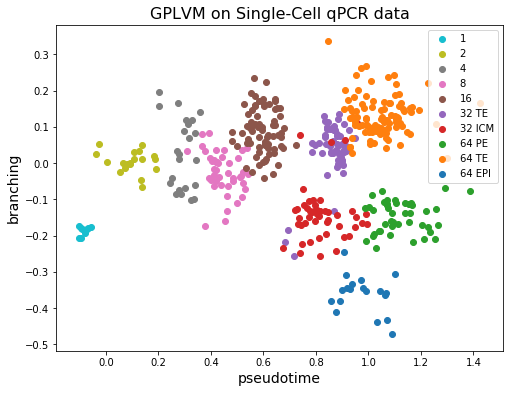
우리는 각 셀에 대한 잠재의 첫 번째 차원(수평축)이 관찰된 캡처 시간(색상)과 잘 일치함을 알 수 있습니다.
반면에 32 TE 셀과 64 TE 셀은 서로 가까이 모여 있습니다. 그리고 ICM 세포가 PE와 EPI로 분화한다는 사실 또한 그림에서 관찰할 수 있습니다!
정리
여기서는 48개의 타겟에 대해서 잠재 변수 2개를 정하는데, 그때 정하는 기준도 시간 정보를 이용하여 사전 분포를 가정하고 진행했습니다.
그래서 학습을 통해 잠재 변수를 학습하고, 그것을 통해 시각화를 2차원으로 해보니, 실제 타겟이 어느 정도 유의미하게 나뉜 것을 알 수 있습니다.
GPLVM은 다음과 같은 가우시안 프로세스를 이용하여 잠재 변수를 발굴하는 알고리즘입니다.
'분석 Python > Pyro' 카테고리의 다른 글
| [Pyro] Application - 5. GP Bayesian Optimization (0) | 2022.08.29 |
|---|---|
| [Pyro] Application - 3. Gaussian Process 이해하기 (0) | 2022.08.28 |
| [Pyro] Application - 2. Bayesian Regression 이해하기 2 (0) | 2022.08.28 |
| [Pyro] Application - 1. Bayesian Regression 이해하기 (1) | 2022.08.21 |
| [Pyro] 개념 파악 및 실습으로 알아보기 (1) | 2022.08.20 |