728x90

광고 한 번만 눌러주세요 블로그 운영에 큰 힘이 됩니다!
R에서는 ggplot이라는 함수를 사용하면 쉽게 group별로 비율 막대그래프를 시각화한다.
그러나 Python은 R과 다르게 Category 변수에 대해서 비율 시각화를 하는 것을 따로 제공하지 않는 것 같다.
## R
ggplot(mtcars,aes(x=factor(cyl),fill=factor(gear)))+
geom_bar(position="fill")
그래서 이번에는 저번에 만들었던 비율 막대 그래포와 Density Plot을 같이 그려보는 것을 하였다.
하는 방법은 위에 R처럼 group별로 하기 위해서 pandas에서 groupby를 사용하고
그다음에 group별로 value_counts로 비율을 계산한 후 바로 bar plot으로 그리면 된다.
aa.groupby('default payment next month')['Category'].value_counts(normalize=True).unstack().plot(kind="bar",stacked= False )

왼쪽은 aa의 데이터 셋이다. 오른쪽 그림은 aa에 있는 dataframe을 Category별로 나눠서 비율을 계산하고 그것을
'default payment next month'별로 시각화한 그림이다.
아래 코드는 각 변수별로 어떤 것은 density plot 어떤 것은 비율 plot을 그린 코드다.
fig , axes = plt.subplots(nrows=4,ncols=6,
figsize=(18,10) )
plt.subplots_adjust(left=0.05, bottom=0.01, right=0.99,
top=0.89, wspace=None, hspace=0.2)
ax = axes.flatten()
sns.set()
for idx , col in enumerate(usecolumn) :
if col in FAC :
tr = data2[col].reset_index(drop=True).values.tolist()
te = gene_data[col].reset_index(drop=True).values.tolist()
if col == "SEX" :
tr = np.array(tr)-1
tr = tr.tolist()
a = tr + te
aa = pd.DataFrame({"Category" :a , col : ["real"] *len(tr) + ["fake"] *len(te) })
aa.groupby(col)['Category'].value_counts(normalize=True).unstack().plot(kind="bar",
stacked= False ,
ax = ax[idx], rot=0)
legend = ax[idx].legend(loc='upper center', bbox_to_anchor=(0.5, 1.0) ,
fontsize=6 , ncol=2,
frameon=False)
#ax[idx].set_xticklabels([])
else :
sns.distplot(data2[col], label = "real" , ax= ax[idx], kde=True)
sns.distplot(gene_data[col], label = "fake" , ax= ax[idx] , kde=True)
ax[idx].set_xticklabels([])
legend = ax[idx].legend(loc='upper center',
bbox_to_anchor=(0.5, 1.0) ,
fontsize=10 , ncol=2,
frameon=False)
frame = legend.get_frame()
frame.set_linewidth(0.0)
frame.set_facecolor('none')
ax[idx].set_title(col,fontsize=15)
#ax[idx].legend(loc='upper center', bbox_to_anchor=(0.5, 1.0) , fontsize=10 , ncol=2)
ax[idx].set_xlabel("")
plt.show()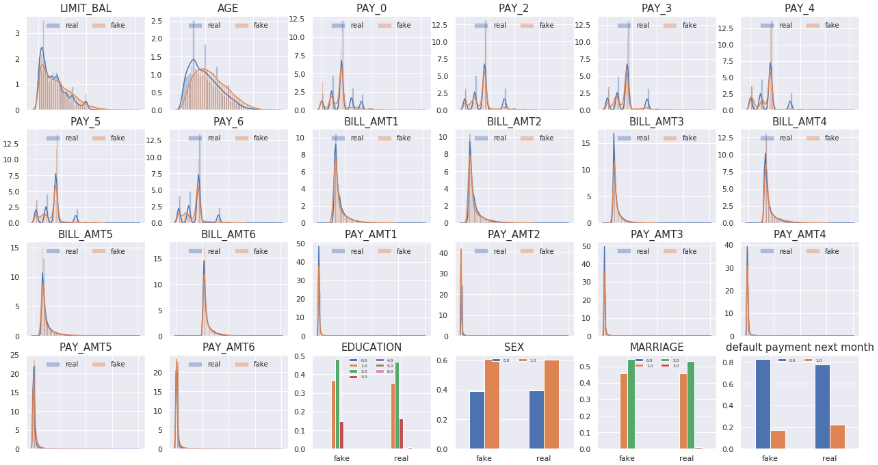
def visualization_two_dataset(table_1 , table_2 , usecolumns , col_n , categorical_var ) :
total_cols = col_n
total_rows = len(usecolumns)//total_cols + 1
fig , axes = plt.subplots(nrows=total_rows,ncols=total_cols,
figsize=(7*total_cols, 7*total_rows), constrained_layout=True)
plt.subplots_adjust(left=0.05, bottom=0.01, right=0.99,
top=0.89, wspace=None, hspace=0.2)
sns.set()
for idx , col in enumerate(usecolumns) :
row = idx//total_cols
pos = idx % total_cols
if col in categorical_var :
table_1_list = table_1[col].reset_index(drop=True).values.tolist()
table_2_list = table_2[col].reset_index(drop=True).values.tolist()
concat = table_1_list + table_2_list
concat_pd = pd.DataFrame({"Category" :concat ,
col : ["table_1"] *len(table_1_list) + ["table_2"] *len(table_2_list) })
concat_pd.groupby(col)['Category'].\
value_counts(normalize=True).unstack().plot(kind="bar",
stacked= False , ax = axes[row][pos] ,rot=0)
legend = axes[row][pos].legend(loc='upper center', bbox_to_anchor=(0.5, 1.0) ,
fontsize=6 , ncol=2,
frameon=False)
else :
sns.distplot(table_1[col].dropna(), label = "real" , ax= axes[row][pos], kde=True)
sns.distplot(table_2[col].dropna(), label = "fake" , ax= axes[row][pos] , kde=True)
axes[row][pos].set_xticklabels([])
legend = axes[row][pos].legend(loc='upper center',
bbox_to_anchor=(0.5, 1.0) ,
fontsize=10 , ncol=2,
frameon=False)
frame = legend.get_frame()
frame.set_linewidth(0.0)
frame.set_facecolor('none')
axes[row][pos].set_title(col,fontsize=15)
axes[row][pos].set_xlabel("")
if total_rows * total_cols > len(usecolumns) :
remainder_n = total_rows * total_cols - len(usecolumns)
for i in range(remainder_n) :
axes[row][-(i+1)].axis("off")
plt.show()'분석 Python > Visualization' 카테고리의 다른 글
| 파이썬 subplots 좀 더 잘 사용해보기 (0) | 2020.03.29 |
|---|---|
| [ Python ] (범례 순서 변경) change legend order (0) | 2020.02.06 |
| [ Python ] 유용한 시각화 함수들 모음 (boxplot, scatter plot, plotly.express, etc) (0) | 2020.01.14 |
| [ Python ] 시각화 여러 개의 그래프 형태 - 1 (0) | 2020.01.12 |
| [ Python ] 이미지들을 동영상으로 만들기 (images -> mp4) (0) | 2020.01.11 |
