728x90
파이썬에서 Sklearn Pipeline 같은 경우 여러 개의 조합을 파이프라인을 만들어서 쉽게 돌릴 수가 있다.
그러나 돌릴 수 있는 것들은 그 모듈의 아규먼트 중에서 fit, transform을 가지고 있는 경우만 가능하다.
차원 축소 방법으로 TSNE, MDS, SpectralEmbedding을 적용하고자 했을 때 아쉽게도 Method가 transform이 없어서 적용할 수가 없다.
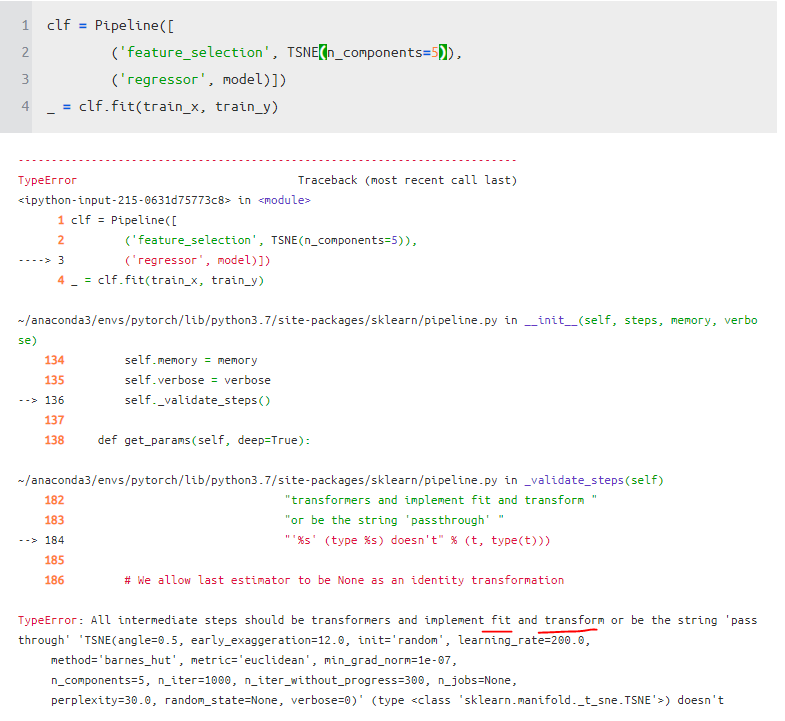

그래서 Sklearn estimator를 새로 만들어서 돌아갈 수는 있게 만든 것을 공유한다.
아래처럼 BaseEstimator와 TransformerMixin을 상속해주면(?) 가능하다
그리고 transform 에는 fit_transform을 써주면 된다.
from sklearn.manifold import (
LocallyLinearEmbedding , TSNE ,
Isomap, SpectralEmbedding, MDS
)
from sklearn.base import BaseEstimator, TransformerMixin
import umap
class TSNEHandler(BaseEstimator, TransformerMixin):
def __init__(self,n_components=2) :
self.tsne =TSNE(n_components=n_components,method="exact")
def fit(self,x) :
self.tsne.fit(x)
def transform(self,x) :
x = self.tsne.fit_transform(x)
return x
def fit_transform(self,x,y) :
x = self.tsne.fit_transform(x)
return x
class MDSHandler(BaseEstimator, TransformerMixin):
def __init__(self,n_components=2) :
self.mds =MDS(n_components=n_components,)
def fit(self,x) :
self.mds.fit(x)
def transform(self,x) :
x = self.mds.fit_transform(x)
return x
def fit_transform(self,x,y) :
x = self.mds.fit_transform(x)
return x
class SpectralEmbeddingHandler(BaseEstimator, TransformerMixin):
def __init__(self,n_components=2,affinity="nearest_neighbors") :
self.Semb =SpectralEmbedding(n_components=n_components,
affinity=affinity)
def fit(self,x) :
self.Semb.fit(x)
def transform(self,x) :
x = self.Semb.fit_transform(x)
return x
def fit_transform(self,x,y) :
x = self.Semb.fit_transform(x)
return x이런 식으로 하면 잘 작동하는 것을 확인했다!
from sklearn.pipeline import Pipeline
model = MLPRegressor(hidden_layer_sizes=(50,20,10),
max_iter= 10000, early_stopping=True ,
learning_rate_init=0.001)
clf = Pipeline([
('dimesion_reduction',TSNEHandler(n_components=5)),
('regressor', model)])
_ = clf.fit(train_x, train_y)
clf = Pipeline([
('dimesion_reduction',MDSHandler(n_components=5)),
('regressor', model)])
_ = clf.fit(train_x, train_y)
clf = Pipeline([
('dimesion_reduction',SpectralEmbeddingHandler(n_components=5)),
('regressor', model)])
_ = clf.fit(train_x, train_y)실제로 돌리고 나서 결과는 아쉬웠다. 아무래도 fit을 데이터마다 새롭게 해 주기 때문에, 데이터의 형태가 동일하다는 가정이 확실하지 않은 한, train과 valid에 성능 차이가 발생하기 때문이다.
'분석 Python > Scikit Learn (싸이킷런)' 카테고리의 다른 글
| 개선된 OneHotEncoder 알아보기(v1.1 이후) (0) | 2022.05.25 |
|---|---|
| [TIP / Sklearn ] Custom Estimator (Ex : Combined Regressor) (0) | 2020.10.27 |
| scikit-learn 파이프라인 시각화 기능 사용 및 재사용 (pipeline visualization) (0) | 2020.05.15 |
| scikit-learn 0.23 이 되면서 변한 점 (0) | 2020.05.15 |
| Scikit-learn Custom Pipeline Save & Reload (저장 및 재사용) (0) | 2020.02.28 |