2020. 1. 15. 00:20ㆍ분석 Python/구현 및 자료
더 빠르게 돌리는 방법 중 하나인 numba에 대한 글에 있는 코드
https://towardsdatascience.com/numba-weapon-of-mass-optimization-43cdeb76c7da
Numba: “weapon of mass optimization”
Numba is a Python compiler, specifically for numerical functions and allows you to accelerate your applications with high performance…
towardsdatascience.com
What is Numba?
Numba is a compiler that allows you to accelerate Python code (numerical functions) for both CPU and GPU:
- Function Compiler: Numba compiles Python functions, not whole applications or parts of it. Basically, Numba is another Python module to improve the performance of our functions.
- Just-in-time: (Dynamic translation) Numba translates the bytecode (intermediate code more abstract than the machine code) to machine code immediately before its execution to improve the execution speed.
- Numerically-focused: Numba is focused on numerical data, such as int, float, complex. For now, there are limitations to use it with string data.
Numba is not the only way to program in CUDA, it is usually programmed in C/C ++ directly for it. But Numba allows you to program directly in Python and optimize it for both CPU and GPU with few changes in our code. In relation to Python, there are other alternatives such as pyCUDA, here is a comparison between them:
CUDA C/C++:
- It is the most common and flexible way to program in CUDA
- Accelerate applications in C, C ++.
pyCUDA
- It is the most efficient CUDA form for Python
- It requires programming C in our Python code and, in general, many code modifications.
Numba
- Less efficient than pyCUDA
- It allows you to write your code in Python and optimize it with few modifications
- It also optimizes the Python code for CPU
Goals
The objectives of this talk are the following:
- Use Numba to compile functions on the CPU
- Understand how Numba works
- Accelerate Numpy ufuncs in GPU
- Write Kernels using Numba (Next tutorial)
from numba import jit
import numpy as np
import math
@jit
def hypot(x, y):
return math.sqrt(x*x + y*y)
# Numba function
hypot(3.0, 4.0)
# Python function
hypot.py_func(3.0, 4.0)
# Python function
%timeit hypot.py_func(3.0, 4.0)
# Numba function
%timeit hypot(3.0, 4.0)
# math function
%timeit math.hypot(3.0, 4.0)
실제 결과를 보면 numba를 사용한 것보다 math.hyopt를 쓴게 더 좋다.
Numba가 각각의 함수 호출에 일정한 오버헤드를 도입하기 때문인데, 이것은 Python 함수 호출 오버헤드보다 더 크고, 매우 빠른 기능이다.
그러나 다른 것에서 Numba 함수를 호출하면 오버헤드가 거의 없으며, 컴파일러를 다른 것과 통합하면 0도 된다. 간단히 말해서, Numba로 기능이 정말로 가속되고 있는지 확인한다
https://numba.pydata.org/numba-doc/dev/user/performance-tips.html#performance-tips
1.14. Performance Tips — Numba 0.48.0.dev0+182.gef119bc-py3.7-linux-x86_64.egg documentation
1.14. Performance Tips This is a short guide to features present in Numba that can help with obtaining the best performance from code. Two examples are used, both are entirely contrived and exist purely for pedagogical reasons to motivate discussion. The f
numba.pydata.org
Numba 작동 방식은 어떻게 될까?

- IR Intermediate Representations
- Bytecode Analysis Intermediate code more abstract than machine code
- LLVM Low Level Virtual Machine, infrastructure to develop compilers
- NVVM It is an IR compiler based on LLVM, it is designed to represent GPU kernels
파이썬의 각 라인에는 여러 줄의 Numba IR 코드가 선행한다.
. inspect_types() np.sin을 그냥 쓰면 할 수 없는 프로세스를 조사할 수 있다.
@jit
def foo_np(x):
return np.sin(x)
foo_np(2)
foo_np.inspect_types()
Creating fractals
# With Numba
from matplotlib.pylab import imshow, ion
@jit
def mandel(x, y, max_iters):
"""
Given the real and imaginary parts of a complex number,
determine if it is a candidate for membership in the Mandelbrot
set given a fixed number of iterations.
"""
i = 0
c = complex(x,y)
z = 0.0j
for i in range(max_iters):
z = z*z + c
if (z.real*z.real + z.imag*z.imag) >= 4:
return i
return 255
@jit
def create_fractal(min_x, max_x, min_y, max_y, image, iters):
height = image.shape[0]
width = image.shape[1]
pixel_size_x = (max_x - min_x) / width
pixel_size_y = (max_y - min_y) / height
for x in range(width):
real = min_x + x * pixel_size_x
for y in range(height):
imag = min_y + y * pixel_size_y
color = mandel(real, imag, iters)
image[y, x] = color
return image
image = np.zeros((500 * 2, 750 * 2), dtype=np.uint8)
%timeit create_fractal(-2.0, 1.0, -1.0, 1.0, image, 20)
img = create_fractal(-2.0, 1.0, -1.0, 1.0, image, 20)
imshow(img)
view rawmandelbrot_numba.py hosted with ❤ by GitHubjit 쓰고 안쓰고의 차이는 다음과 같다.
4.62 seconds -> 52.4ms 그냥 함수에다가 jit을 쓰면 돼서 참 신기한 것 같다.
Numba는 오직 연속형 함수에서만 작동한다고 했다. 비록 어떤 파이썬 코드에서도 Numba compile과 작동을 하지만,
아직 컴파일할 수 없는(사전과 같은) 종류의 데이터도 있고, 컴파일하는 것도 말이 안 된다고 말해 왔다.
@jit
def dictionary(dict_test):
return dict_test['house']
dictionary({'house': 2, 'car': 35})그러나 이것은 지금 실패하지 않았다! Numba는 ditionaries를 compile하지 못한다.
핵심은 Numba는 2가지 함수들을 만든다는 것이다.
하나는 Python 또다른 하나는 Numba이다.
이것을 증명하는 방법은 nopython=True를 하면 아마 Numba만 작동하게 하는 것 같다.
그다음에 dictionary를 해보면 결과는 다음과 같다.
@jit(nopython = True)
def dictionary(dict_test):
return dict_test['house']
dictionary({'house': 2, 'car': 35})
Numba for GPU
1. ufuncs/gufuncs__
2. CUDA Python Kernels (Next tutorial)
@vertorize 함수를 이용하여 CPI에 최적화하고 compile 할 수 있다.
from numba import vectorize
a = np.array([1, 2, 3, 4])
b = np.array([23, 341, 12, 5])
@vectorize
def add_ufunc_cpu(a, b):
return a + b
# Numba function
add_ufunc_cpu(a, b)이전 함수를 시행하고 compile 하기 위해서 cpu를 사용하는 대신에 CUDA를 사용할 수 있다.
사용하기 위해서는 target attribute를 사용해야 한다.
그리고 type을 구체적으로 정해준다.
@vectorize(['int64(int64, int64)'], target='cuda')
def add_ufunc_gpu(x, y):
return x + y
add_ufunc_gpu(a, b)
기대했던 결과랑은 달리 np.add가 훨씬 좋다!!
왜 이런지에 대해서 알려면, Numba는 어떻게 하는지 알아야 한다.
- Compile a CUDA kernel to execute the ufunc function in parallel over all the elements of the input array
- Assign the inputs and outputs to the GPU memory
- Copy the input to the GPU
- Run the CUDA Kernel
- Copy the results back from the GPU to the CPU
- Return the results as a numpy array
C의 구현에 비해 Numba는 이러한 유형의 작업을 보다 간결하게 수행할 수 있도록 한다.
왜 CPU보다 GPU가 더 느릴까?
- 인풋들이 매우 작다.
GPU는 수천건의 값들에 대해서 한 번에 병렬로 함으로써 더 좋은 성능을 낸다.
그러나 예제에서는 그렇지 않았다. 그래서 큰 행렬에서는 GPU가 훨씬 나을 것이다. - 매우 간단한 계산이였다.
계산을 GPU로 전송하려면 CPU 기능을 호출하는 것에 비해 많은 "effort"가 필요하다.
과도한 계산이 있다면 CPU보다는 GPU가 더 빠를 것이다. - NUMBA는 CPU에서 데이터를 복사한다.
- 변수 타입이 필요 이상으로 크다.
현재 int64를 사용하고 있지만, 실제로 cpu에서는 32 ,64 비트가 동일한 처리 속도를 가지지만,
GPU에서는 64bit가 32bit보다 24배 느리다고 한다.
그래서 실제로 gpu 함수에선 이런 변수 타입이 굉장히 중요하다.
이것을 염두에 두고, 이전 포인트에서 배운 것을 적용하여, CPU보다 GPU에서 운용하는 것이 정말 빠른지 알아보려고 노력해보면 다음과 같다.
import math
sqrt_pi = np.float32((2*math.pi)**0.5)
@vectorize(['float32(float32, float32, float32)'], target='cuda')
def gaussian_dens_gpu(x, mean, sigma):
return math.exp(-0.5 * ((x - mean) / sigma)**2) / (sigma * sqrt_pi)
x = np.random.uniform(-3, 3, size=1000000).astype(np.float32)
mean = np.float32(0.0)
sigma = np.float32(1.0)
# We use scipy to perform the same calculation but on the CPU and compare it with the GPU
import scipy.stats
norm_pdf = scipy.stats.norm

import math
sqrt_pi = np.float32((2*math.pi)**0.5)
@vectorize
def gaussian_dens_cpu(x, mean, sigma):
return math.exp(-0.5 * ((x - mean) / sigma)**2) / (sigma * sqrt_pi)
x = np.random.uniform(-3, 3, size=1000000).astype(np.float32)
mean = np.float32(0.0)
sigma = np.float32(1.0)
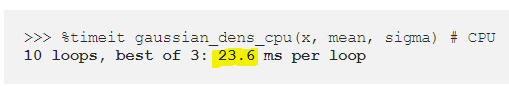
%timeit gaussian_dens_cpu(x, mean, sigma) # CPU불행히도, ufunch의 정의 범위에 속하지 않는 몇 가지 기능이 있으므로, 우리가 cuda.jit를 사용하는 요건을 충족하지 않는 GPU의 기능을 실행한다. 우리는 GPU에서 실행되는 "장치 기능"을 사용할 수 있다.
from numba import cuda
import numpy as np
import math
from numba import vectorize
# Device function
@cuda.jit(device=True)
def polar_to_cartesian(rho, theta):
x = rho * math.cos(theta)
y = rho * math.sin(theta)
return x, y
@vectorize(['float32(float32, float32, float32, float32)'], target='cuda')
def polar_distance(rho1, theta1, rho2, theta2):
x1, y1 = polar_to_cartesian(rho1, theta1)
x2, y2 = polar_to_cartesian(rho2, theta2)
return ((x1 - x2)**2 + (y1 - y2)**2)**0.5
n = 1000000
rho1 = np.random.uniform(0.5, 1.5, size=n).astype(np.float32)
theta1 = np.random.uniform(-np.pi, np.pi, size=n).astype(np.float32)
rho2 = np.random.uniform(0.5, 1.5, size=n).astype(np.float32)
theta2 = np.random.uniform(-np.pi, np.pi, size=n).astype(np.float32)
import os
os.environ["CUDA_VISIBLE_DEVICES"]="1"
%timeit polar_distance(rho1, theta1, rho2, theta2)
아주 쉽게 쓸 수 있는 것 같고 빨리 하기 위해서 요즘 ray도 있는 것 같은데 같이 알아봐야겠다!
https://numba.pydata.org/numba-doc/dev/cuda/index.html
3. Numba for CUDA GPUs — Numba 0.48.0.dev0+182.gef119bc-py3.7-linux-x86_64.egg documentation
numba.pydata.org
'분석 Python > 구현 및 자료' 카테고리의 다른 글
| [ Python ] Install all dependency packages (0) | 2020.01.25 |
|---|---|
| [ Python ] Optuna Sampler 비교 (TPESampler VS SkoptSampler) (0) | 2020.01.24 |
| [ Python ] smtplib을 활용한 메일 보내기 (이미지 , 텍스트 , gif) (0) | 2020.01.05 |
| [ Python ] 동적 변수 할당 및 불러들이기 (1) | 2019.12.15 |
| [ Python ] H2O XGBOOST Practice (0) | 2019.12.15 |