728x90
This is because when a process requests for memory, memory is allocated in two ways:
- Contiguous Memory Allocation (consecutive blocks are assigned)
- NonContiguous Memory Allocation(separate blocks at different locations)
Pandas는 RAM에 데이터를 적재하기 위해서 Contiguous Memory 방식을 사용한다.
왜냐하면 읽고 쓰는 것은 디스크보다 RAM에서 하는 것이 더 빠르게 때문이다.
- Reading from SSDs: ~16,000 nanoseconds
- Reading from RAM: ~100 nanoseconds
몇 가지 팁들이 읽길래 공유한다.
import pandas as pd
import warnings
warnings.filterwarnings("ignore")
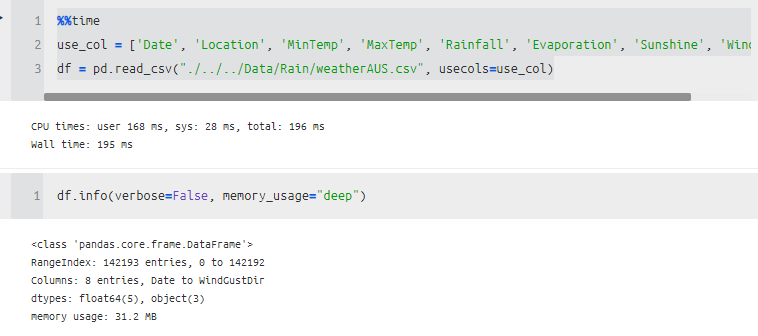
1. usecols
원하는 것만 뽑아서 사용하는 방법이다.


2. Using correct dtypes for numerical data or object :
- int8 can store integers from -128 to 127.
- int16 can store integers from -32768 to 32767.
- int64 can store integers from -9223372036854775808 to 9223372036854775807.
WindGustDir 같은 경우 object를 category로 바꿔주니 메모리가 많이 줄어든 것을 알 수 있다.
그래서 미리 타입만 알고 있다면, 쉽게 데이터를 빠르게 읽을 수 있다.
%%time
df = pd.read_csv("./../../Data/Rain/weatherAUS.csv",
usecols=use_col,
dtype={"WindGustDir": "category"})

%%time
df = pd.read_csv("./../../Data/Rain/weatherAUS.csv",
usecols=use_col,
dtype={"WindGustDir": "category",
"Evaporation" : "float16",
})
꿀팁 : missing value 또는 결측이 많은 경우 Sparse Series로 변경하여 메모리 감소가 가능하다.
series = df['Evaporation']
series.memory_usage(index=False, deep=True)
## 1137544
sparse_series = series.astype("Sparse[str]")
sparse_series.memory_usage(index=False, deep=True)
## 4388832?? 줄어드는 거 맞나
꿀팁 결측치를 사전에 원하는 값으로 변경하면 메모리 감소
%%time
def convertor(val) :
if val == np.nan :
return -9999
return val
df = pd.read_csv("./../../Data/Rain/weatherAUS.csv",
usecols=use_col,
converters={"Evaporation": convertor},
dtype={"WindGustDir": "category",
"Evaporation" : "float16",
})
3. nrows, skip rows
df = pd.read_csv("./../../Data/Rain/weatherAUS.csv", usecols=use_col, nrows =100 , skip_na=[0,2,5])
%%time
sample = pd.read_csv("./../../Data/Rain/weatherAUS.csv")
dtypes = df.dtypes # Get the dtypes
cols = df.columns # Get the columns
dtype_dictionary = {}
for c in cols:
"""
Write your own dtypes using
# rule 2
# rule 3
"""
if str(dtypes[c]) == 'int64':
dtype_dictionary[c] = 'float32' # Handle NANs in int columns
elif str(dtypes[c]) == 'object':
dtype_dictionary[c] = 'category'
else:
dtype_dictionary[c] = str(dtypes[c])
dtype_dictionary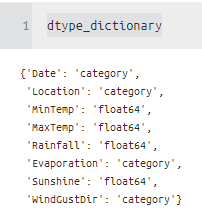
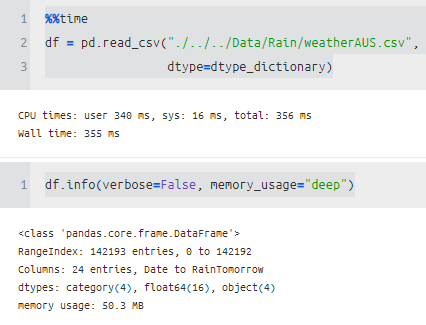
%%time
df = pd.read_csv("./../../Data/Rain/weatherAUS.csv",
dtype=dtype_dictionary)
##CPU times: user 340 ms, sys: 16 ms, total: 356 ms
##Wall time: 355 ms
4. Loading Data in Chunks:
df = pd.read_csv("train.csv", chunksize=1000)
total_len = 0
for chunk in df:
# Do some preprocessing to reduce the memory size of each chunk
total_len += len(chunk)
tp = pd.read_csv('train.csv', iterator=True, chunksize=1000) # gives TextFileReader
df = pd.concat(tp, ignore_index=True)
%%time
tp = pd.read_csv("./../../Data/Rain/weatherAUS.csv",dtype=dtype_dictionary, chunksize=1000)
df = pd.concat(tp,ignore_index=True)
df.shape
# (142193, 24)5. Multiprocessing using pandas:
%%time
LARGE_FILE = "./../../Data/Rain/weatherAUS.csv"
CHUNKSIZE = 1000 # processing 100,000 rows at a time
def process_frame(df):
# process data frame
return df
if __name__ == '__main__':
reader = pd.read_csv(LARGE_FILE, chunksize=CHUNKSIZE,dtype=dtype_dictionary)
pool = mp.Pool(4) # use 4 processes
funclist = []
for df in reader:
# process each data frame
f = pool.apply_async(process_frame,[df])
funclist.append(f)
df = pd.concat([f.get() for f in funclist],axis=0)6. Dask Instead of Pandas:
import dask.dataframe as dd
data = dd.read_csv("./../../Data/Rain/weatherAUS.csv",
dtype=dtype_dictionary,
assume_missing=True)
data.compute()
⚡️ Load the same CSV file 10X times faster and with 10X less memory⚡️
Pandas, Dask, MultiProcessing, Etc…
towardsdatascience.com
'분석 Python > Pandas Tip' 카테고리의 다른 글
| [TIP / Pandas] Change Columns Order (열 순서 바꾸기) (0) | 2020.10.28 |
|---|---|
| [Python] Pandas를 활용하여 엑셀 시트별로 만들기 (0) | 2020.10.20 |
| [Pandas] 조건걸고 새로운 컬럼 추가하기 (1) | 2020.08.12 |
| [Pandas] data type별로 컬럼들을 사전 형태로 모으기 (0) | 2020.07.23 |
| [Pandas] 여러개의 컬럼 하나로 합치기 (0) | 2020.07.22 |